BBC News, 11 décembre 2022
Édition ciblée d'une base du génome
une thérapie révolutionnaire éradique le cancer incurable d'une fille de 13 ans
par James Gallagher, Correspondant Science et santé
Le cancer incurable d'une adolescente a été éliminé de son corps lors de la première utilisation d'un nouveau type de traitement révolutionnaire. Tous les autres traitements de la leucémie d'Alyssa avaient échoué. Ainsi, les médecins de l'hôpital Great Ormond Street ont utilisé "l'édition de base" pour réaliser un exploit d'ingénierie biologique afin de lui créer un nouveau médicament vivant.
Six mois plus tard, le cancer est indétectable, mais Alyssa est toujours sous surveillance au cas où il reviendrait.

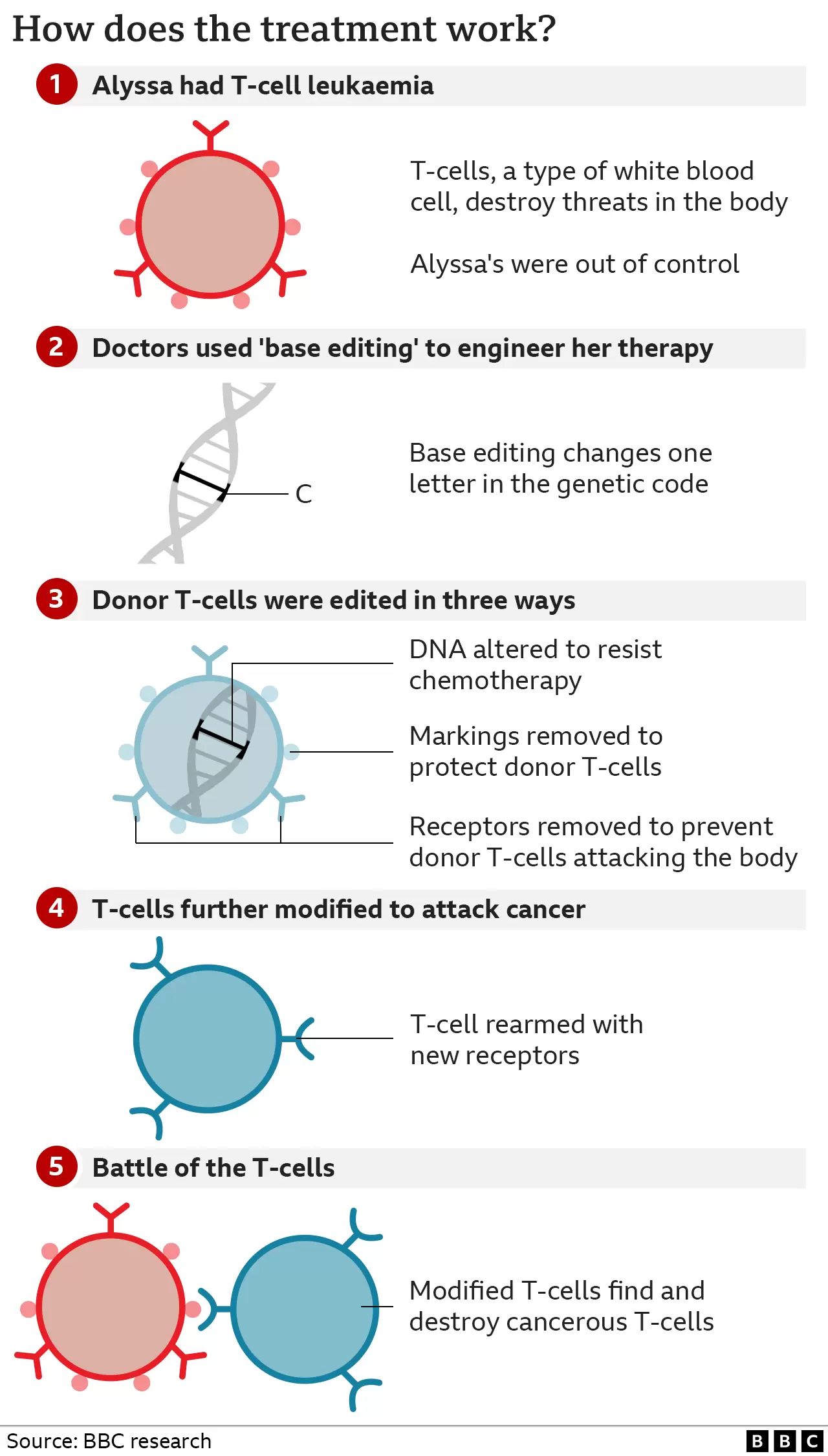
Alyssa, âgée de 13 ans et originaire de Leicester, a reçu un diagnostic de leucémie aiguë lymphoblastique à cellules T (note perso : les cellules du système immunitaire) en mai de l'année dernière.
Les lymphocytes T sont censés être les gardiens du corps - recherchant et détruisant les menaces - mais pour Alyssa, ils étaient devenus le danger et devenaient incontrôlables. Son cancer était agressif. La chimiothérapie, puis une greffe de moelle osseuse, n'ont pas réussi à en débarrasser son corps. Sans la médecine expérimentale, la seule option qui restait aurait été simplement de rendre le reste de la vie d’Alyssa aussi confortable que possible.
"Finalement, je serais décédée", a déclaré Alyssa. Sa mère, Kiona, a déclaré à cette époque, l'année dernière, qu'elle redoutait Noël, "pensant que c'était notre dernier avec elle". Et puis elle "a juste pleuré" au 13e anniversaire de sa fille en janvier.
Ce qui s'est passé ensuite aurait été impensable il y a quelques années à peine et a été rendu possible grâce aux progrès incroyables de la génétique. L'équipe de Great Ormond Street a utilisé une technologie appelée édition de base, qui a été inventée il y a seulement six ans (voir ci-dessous). Les bases sont le langage de la vie. Les quatre types de base - adénine (A), cytosine (C), guanine (G) et thymine (T) - sont les éléments constitutifs de notre code génétique. Tout comme les lettres de l'alphabet énoncent des mots porteurs de sens, les milliards de bases de notre ADN énoncent le manuel d'instructions de notre corps.
L'édition de base permet aux scientifiques de zoomer sur une partie précise du code génétique, puis de modifier la structure moléculaire d'une seule base, en la convertissant en une autre et en modifiant les instructions génétiques. Une équipe nombreuse de médecins et de scientifiques a utilisé cet outil pour concevoir un nouveau type de cellule T capable de traquer et de tuer les cellules T cancéreuses d'Alyssa.
Ils ont commencé avec des lymphocytes T sains provenant d'un donneur et se sont mis à les modifier.
La première modification de base a désactivé le mécanisme de détection propre aux lymphocytes T, afin qu'ils n'agressent pas le corps d'Alyssa
Le second a supprimé un marquage moléculaire, appelé CD7, qui se trouve sur toutes les cellules T.
La troisième modification était une cape d'invisibilité qui empêchait les cellules d'être tuées par un médicament de chimiothérapie.
La dernière étape de la modification génétique a demandé aux lymphocytes T d'aller à la recherche de tout ce qui portait le marquage CD7 afin qu'ils détruisent tous les lymphocytes T de son corps, y compris les cellules cancéreuses. C'est pourquoi ce marquage doit être retiré de la thérapie - sinon elle se détruirait elle-même.
Si la thérapie fonctionne, le système immunitaire d'Alyssa - y compris les lymphocytes T - sera reconstruit avec la deuxième greffe de moelle osseuse.
Lorsque l'idée a été expliquée à la famille, sa mère Kiona s'est dit : « Vous pouvez faire ça ? ». Alyssa pris la décision d'être la première à suivre la thérapie expérimentale - qui contenait des millions de cellules modifiées - en mai de cette année.
"Elle est la première patiente à être traitée avec cette technologie", a déclaré le professeur Waseem Qasim, du Great Ormond Street Institute of Child Health (ICH) de l’University College de Londres (UCL).
Il a déclaré que cette manipulation génétique était un "domaine scientifique en évolution très rapide" avec un "potentiel énorme" dans toute une gamme de maladies.
Alyssa est restée vulnérable à l'infection, car les cellules de synthèse attaquaient à la fois les cellules T cancéreuses de son corps et celles qui la protégeaient de la maladie. Après un mois, Alyssa était en rémission et a reçu une deuxième greffe de moelle osseuse pour faire repousser son système immunitaire.
Alyssa a passé 16 semaines à l'hôpital et n'a pas pu voir son frère, qui allait encore à l'école, au cas où il apporterait des infections. On s’est inquiété après que le contrôle de trois mois ait trouvé à nouveau des signes de cancer. Mais ses deux contrôles les plus récentes ont été bons.
"Vous apprenez simplement à apprécier chaque petite chose. Je suis tellement reconnaissante d'être ici maintenant", a déclaré Alyssa. "C'est fou. C'est juste incroyable que j'aie pu avoir cette opportunité, j'en suis très reconnaissante et ça va aussi aider d'autres enfants à l’avenir. »
Elle apprécie Noël, est demoiselle d'honneur au mariage de sa tante, remonte sur son vélo, retourne à l'école et "fait juste des trucs de gens normaux". La famille espère que le cancer ne reviendra jamais, mais est déjà reconnaissante du temps qu'il leur a fait gagner. "Avoir cette année supplémentaire, ces trois derniers mois quand elle est restée à la maison, a été un cadeau en soi", a déclaré sa mèreKiona. Son père James a déclaré: "Je trouve assez difficile de dire à quel point nous sommes fiers. Quand vous voyez ce qu'elle a traversé et sla vitalité dont elle a fait preuve à chaque situation, c'est exceptionnel. »
La plupart des enfants atteints de leucémie répondent aux principaux traitements, mais on pense que jusqu'à une douzaine par an pourraient bénéficier de cette thérapie.
Alyssa n'est que la première des 10 personnes à recevoir ce traitement dans le cadre d'un essai clinique. Le Dr Robert Chiesa, du service de greffe de moelle osseuse du Great Ormond Street Hospital, a déclaré : « C'est extrêmement excitant. De toute évidence, il s'agit d'un nouveau domaine de la médecine et il est fascinant de pouvoir rediriger le système immunitaire pour lutter contre le cancer. La technologie, cependant, ne fait qu'effleurer la surface de ce que l'édition de base pourrait réaliser. Le Dr David Liu, l'un des inventeurs de l'édition de base à l'Université d'Harvard, a déclaré que c'était "un peu surréaliste" que des gens soient traités six ans seulement après l'invention de la technologie.
Dans la thérapie d'Alyssa, chacune des modifications de base impliquait de casser une section du code génétique afin que cela ne fonctionne plus. Mais il existe des applications plus nuancées où, au lieu de désactiver une instruction, vous pouvez en réparer une défectueuse. La drépanocytose, aussi appelée "anémie falciforme", par exemple, est causée par un seul changement de base qui pourrait être corrigé. Il y a donc déjà des essais d'édition de base en cours dans la drépanocytose, ainsi que dans l'hypercholestérolémie famillale et la bêta-thalassémie, un trouble sanguin.
Le Dr Liu a déclaré que "les applications thérapeutiques de l'édition de base ne font que commencer" et qu'il fallait « rester humble du fait d'appartenir cette ère de l'édition thérapeutique des gènes humains", car la science prenait désormais "des mesures clés pour prendre le contrôle de nos génomes".
L'Édition programmable d'une seule base ciblée dans l'ADN du génome, sans coupure de la double hélice. (retour)
En 2012, Jennifer Doudna et Emmanuelle Charpentier publient un article révolutionnaire dans le domaine de la biologie moléculaire. Après avoir élucidé tout le mécanisme de défense immunitaire des bactéries contre les virus bactériophages, elles s'inspirent de ce mécanisme pour créer un toute nouvelle approche permettant d'éditer (= modifier) un gène défectueux à l'aide d'un ARN guide qui est construit pour repérer spécifiquement le gène et l'endroit du gène à modifier, et y amener une enzyme appelée Cas9 capable de couper les deux brins entrelacés de l'ADN. Cette cassure effectuée, il est possible d'en corriger une séquence de nucléotides homologues sur les deux brins, ou au contraire d'effectuer des opérations telles que des délétions de nucléotides pour déactiver l'expression d'un gène qui est traduit en une protéine à caratère pathologique. Cette méthode porte le nom de CRISPR/Cas9.
Cette approche révolutionnaire permettant l'édition de n'importe quel gène du génome, et ouvrant la voie à la correction de maladies génétiques graves pour lesquelles il n'existe aucun traitement, telles que l'anémie falciforme ou la chorée de Huntington, a valu à ces deux chercheuses, le Prix Nobel de Chimie 2020, à peine 8 ans après leur premièrepublication. Nous avons déjà décrit cela au moment où le Prix Nobel leur a été attribué.
Toutefois, la méthode initiale souffrait encore de plusieurs défauts. Ainsi, très souvent, la rupture des deux brins tend à se réparer spontanément par jonctions d'extrémités non-homologues, conduisant à des mutations ou des délétions. Si ce type de mécanisme peut dans certains cas être acceptable quand on veut désactiver un gène déficient, il ne permet pas de réparer un gène déficient pour lui permettre de pouvoir transmettre l'information désirée, au travers de sa traduction en une protéine fonctionnelle. Un autre défaut est le nombre parfois trop élevé de ruptures de la double hélice en des endroits non désirés. On parle alors de coupures hors cible (off-target cleavage).
Depuis, diverses améliorations ont été apportées à la technique par de nombreuses équipes universitaires, dont celle de Jennifer Doudna elle-même. Mais la technique développée par l'équipe du Professeur David R. Liu de l'Université d'Harvard est à mon sens une des plus innovatrices, en ce sens qu'elle s'inspire le le technologie CRISPR/Cas9, mais en s'en écartant d'une part, et en la complétant d'autre part, pour pouvoir éditer une seule base nucléotidique précise du génome, ayant observé que la grande majorité des maladies génétiques sont dues qu'a une mutation ponctuelle d'une seule base d'un gène particulier.
Nous avons déjà vu à plusieurs reprises que l'ADN est constitué de deux hélices entrelacées formées de l'alternance irrégulière de 4 types de bases, appelées nucléotides : l'adénine (A), la thymine (T), la guanine (G) et la cytosine (C). Ces bases sont toutes liées à un sucre identique, le désoxyribose, lequel est lié à un groupement phosphate, lié à son tour à un autre désoxyribose portant lui-même un nucléotide, et ainsi de suite. Cette chaîne se lie à une autre chaîne par des liaisons de faibles énergies, appelées ponts hydrogènes, entre nucléotides de chacune d'elles. Mais cet appariement n'est possible qu'entre une adenine et une thymine ou entre une guanine et une cytosine, si bien que les deux chaines réunies ne comportent entre elles que des liaisons A-T et G-C. Cet appariemment étant effectué, les deux chaines (ou "brins d'ADN") forment spontanément une double hélice entrelacée, l'une gauche, l'autre droite, dont la structure avait été identifiée par Watson et Crick en 1953.
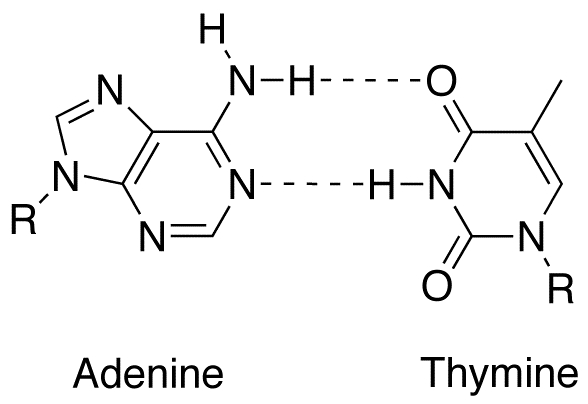
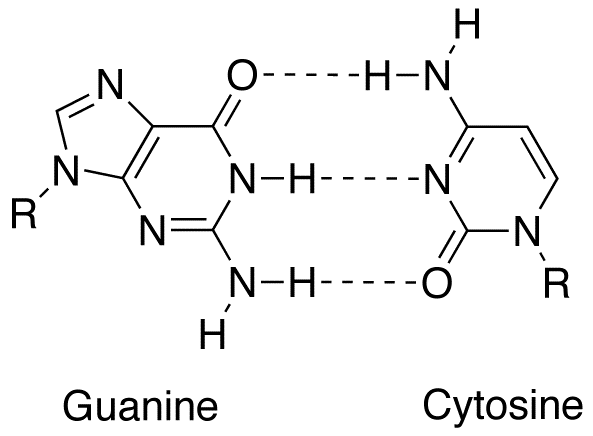
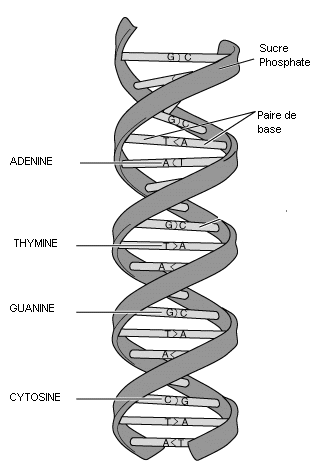
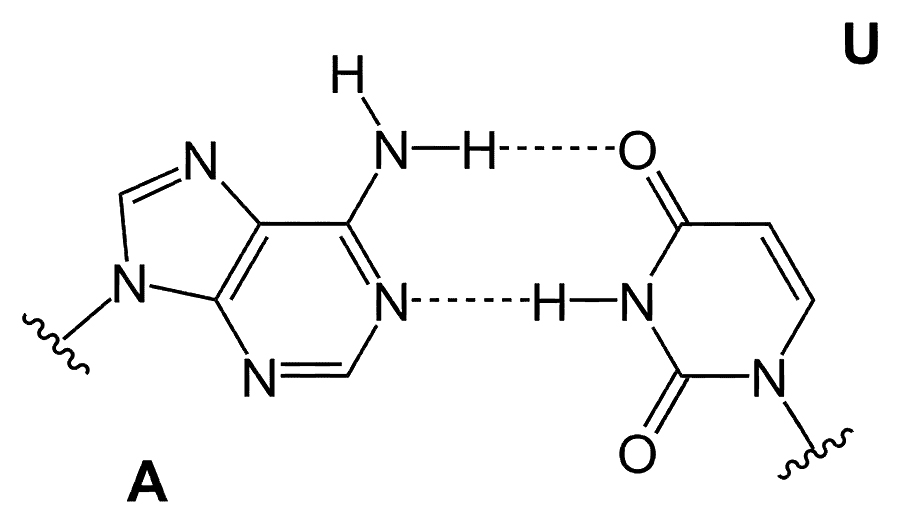
L'équipe de David Liu a modifié la structure de l'enzyme Cas9 de façon à la rendre incapable de couper les deux brins, mais qui conserve la capacité de reconnaître l'emplacement de l'ARN guide fixé à un endroit spécifique de l'ADN par appariemment de bases, ainsi que de rompre les liaisons A-T et G-C sur une certaine distance, formant ainsi une bulle dans laquelle les A et les T ainsi que les G et les C ne sont plus solidaires l'un de l'autre. Il appelle cette Cas9 modifiée dCas9 pour "Catalitycally dead Cas9". Ils ont ensuite construit un assemblage constitué de la dCas9, de l'ARN guide, et d'une enzyme capable d'hydrolyser une amine primaire NH2 d'une cytosine ou celle d'une adénine, à une courte distance de l'endroit où la dCas9 vient se fixer à l'ADN (sur un triplet spécifique de nucléotides appelé PAM (pour "Protospacer Adjacent Motif"), le motif adjacent à ce qui deviendra la bulle dans laquelle les nucleotides des deux brins sont écartés, et ou se fera la rupture des deux brins d'ADN dans le cas de Cas9, mais pas dans le cas de dCas9). À cela, ils ajoutent encore une enzyme appelée nikase capable de couper un seul des deux brins d'ADN, et qu'ils ajustent pour que cette nikase coupe le brin opposé à celui où une cytosine ou une adénine a subi une hydrolyse de son NH2.
L'hydrolyse d'une cytosine produit une autre base appelée uracyle (U), qui a les mêmes propriétés d'appariemment qu'une thymine (T). Par ailleurs, l'hydrolyse d'une adenine produit une inosine (I), laquelle est vue par la polymérase (l'enzyme fabriquant une nouvelle chaîne d'ADN, en tant que copie complémentaire de la précédente, c.a.d. où un T remplacera un A et ou un C remplacera un G) comme une guanine G, pouvant s'apparier à une cytosine C.
Dans le cas de la transformation de C en U, la réparation de l'autre brin sectionné se fera pour restituer les bases complémentaires, mais cette fois, U étant vue comme similaire à T, c'est une adénine A qui sera en face de U, et plus la guanine qui était appariée à C. Et donc, quand ces deux chaines complémentaires d'ADN se répliqueront, pour former deux nouvelles doubles hélices, comme cela se produit à chaque division cellulaire par mitose, là où une mutation d'une seule cytosine avait été provoquée, la liaison C-G se voit donc finalement transformée en liaison T-A dans toutes les nouvelles doubles hélices.
L'équipe de David Liu a commencé par s'attaquer à cette mutation de C, car l'enzyme hydrolysant une cytosine en uracyle existe naturellement dans les outils des acides nucléiques. D'ailleurs, dans l'ARN, la thymine (T) n'est pas présente. Elle y est naturellement substituée par une uracyle (U). Par contre, au complexe qu'ils ont construit, ils ont du également ajouter un inhibiteur de glycosylase de l'uracyle, car sans être glycosylée (c'est à dire protégée par un ensemble de molécules de sucres, l'uracyle est très rapidement reconnue par les enzymes de réparation, et remplacée par la cytosine d'origine. Leurs travaux, qui ont étudiés divers complexes, en vue d'améliorer le rendement de mutation a conduit à de trsès bons rendements et surtout à un très faible taux de mutations off-target. Ces travaux sont repris dans l'article ci-dessous, publié en 2016 :
Komor, A., Kim, Y., Packer, M. et al. Programmable editing of a target base in genomic DNA without double-stranded DNA cleavage. Nature 533, 420–424 (2016).
Ils se sont ensuite consacrés à la mutation de l'autre liaison A-T en G-C. La tâche était moins évidente car il n'existait aucune enzyme capable d'hydrolyser le NH2 de l'ADN dans la boîte à outils de l'ADN. Mais leurs recherches ont fini par permettre d'identifier une enzyme appelée TadA, une adnine deaminase, hydrolysant l'adénine en inosine, dans de petits ARN de transfert (tRNA) utilisés pour amener dans les ribosomes (les machines à fabrication de protéines) les différents types d'acides aminés constitutifs des protéines. En effectuant diverses mutaions dans cette enzyme TadA, ils ont pu la rendre compatible avec la déamination (hydrolyse du NH2) de l'adénine dans l'ADN. L'inosine (I) formée est capable de se lier à une cytosine (C), bien que de façon "non-Watson-Crick", mais n'est pas reconnue par les enzymes de réparation, et quand l'ADN-polymérase générera une nouvelle chaîne complémentaire lors de la réplication qui suivra, cette inosine sera confondue avec une guanine (G). Ils ont donc construit un complexe constitué d'un ARN guide, d'une dCas9, de la déaminase modifiée d'ADN et d'une nikase de la chaîne opposée. L'inosine formée par déamination de l'adénine n'étant pas reconnue par les enzymes de réparation, un inhibiteur de glycosylase n'était pas nécessaire dans ce cas. Ici aussi, après plusieurs optimisations, ils sont arrivés à un très bon rendement de mutation, avec également un très faible taux de mutations off target. Ces travaux ont été publiés l'année suivante, en novembre 2017 :
Gaudelli, N., Komor, A., Rees, H. et al. Programmable base editing of A•T to G•C in genomic DNA without DNA cleavage. Nature 551, 464–471 (2017).
Les deux séries de travaux de l'équipe du Professeur David R. Liu de l'Université d'Harvard permettent donc d'adresser toutes les mutations ponctuelles de nucléotides par cette echnique d'édition de base, puisque, qu'il s'agisse de corriger une mutaion ponctuelle de A, T, G ou C, elle pourra l'être en éditant soit A soit C, puisque la base complémentaire sera de facto également modifiée au travers de la liaison A-T ou G-C après réplication de la double hélice. (retour)