SCIENCE NEWS ( dans Science, Vol 376, Issue 6597 )
1er juin 2022
Mieux que CRISPR ? Une autre façon de résoudre les problèmes génétiques peut être plus sûre et plus polyvalente
L'édition d'épigénome inverse les interrupteurs marche-arrêt génétiques dans les études sur la souris

par Jocelyn Kaiser
du magazine Science
Des outils tels que CRISPR qui coupent l'ADN pour modifier sa séquence se rapprochent de manière attractive de la clinique en tant que traitements de certaines maladies génétiques. Mais loin des feux de la rampe, les chercheurs sont de plus en plus enthousiasmés par une alternative qui laisse une séquence d'ADN inchangée. Ces outils moléculaires ciblent l'épigénome, les étiquettes chimiques ornant l'ADN et ses protéines environnantes qui régissent l'expression d'un gène et son comportement final.
( NOTE PERSONNELLE : l’édition de gènes par la méthode CRISPR-Cas9 a été publiée en 2012 par deux chercheuses, l’une française, Emmanuelle Charpentier, l’autre américaine, Jennifer Doudna. Leurs travaux leur ont valu le Prix Nobel de Chimie en 2020, et cette innovation fut si spectaculaire que je l’ai décrite à l’époque en détail sur mon site :
La méthode décrite ici s’en inspire directement, car elle utilise les travaux de Charpentier et Doudna pour reconnaître le gène à activer ou désactiver, mais ne coupe pas l’ADN du gène en vue de le modifier, mais modifie les protéines avoisinantes qui l’expriment ou le rende silencieux. Ces processus épigénomiques sont essentiels pour différencier une cellule d’un type, d’une cellule d’un autre type, car toutes les cellules de notre corps portent le même génome sous forme d’ADN, et ce sont les différences existantes dans l’épigénome qui les différencient. Les deux agents principaux de l’épigénome sont d’une par des méthylations de certaines bases de l’ADN, qui rendent le gène silencieux, ou des histones, qui sont des protéines autour desquelles les fibres d’ADN s’enroulent, et dont le changement de conformation permet l’expression ou l’inactivation d’un gène. Ces mécanismes sont fondamentaux pour distinguer une cellule d’une autre, un neurone d’une cellule musculaire par exemple, et d’ailleurs, à partir de cellules souches non différenciées, il est possible de générer toutes les cellules différentes d’un individu, par cette différentiation cellulaire.
Mais parfois, l’épigénome commet des erreurs, ou l’ADN subit des mutations rendant l’épigénome inefficace. Dans ce cas, cela peut conduire à des pathologies, parfois très graves, telles que les cancers ou la terrible maladie de Huntington, une maladie autosomique dominante qui conduit à la mort par neurodégénérescence, et qui, étant dominante, c’est à dire qu’il suffit qu’un seul des deux gènes identiques d’une paire de chromosomes soit atteint pour que la personne développe la maladie. Quand un parent est atteint, un enfant a donc 50% de chances d’en être atteint à son tour. C’est là que cette technologie trouve tout son champ d’application. Et comme nous allons le voir, elle tire parti à la fois des travaux de Charpentier et Doudna sur la méthode CRISPR-Cas9, mais aussi des travaux de Katalin Kariko et Drew Weissman sur l’encapsulation dans des nanocapsules de phospholipides d’ARN messagers, travaux qui ont conduit à l’élaboration des vaccins contre le Covid-19
Plus d'info sur l'épigénétique )
Une vague d'études au cours des dernières années chez la souris suggère que l'édition de l'épigénome est un moyen potentiellement plus sûr et plus flexible d'activer ou de désactiver les gènes que l'édition de l'ADN. Dans un exemple décrit le mois dernier lors d'une réunion de thérapie génique à Washington, D.C., une équipe italienne a réduit l'expression d'un gène chez la souris pour abaisser le taux de cholestérol des animaux pendant des mois. D'autres groupes explorent l'édition de l'épigénome pour traiter une grande variété de pathologies, du cancer à la douleur en passant par la maladie de Huntington, un trouble cérébral mortel.
Contrairement à l'édition de l'ADN, où les modifications sont permanentes et peuvent inclure des résultats imprévus, les modifications épigénomiques peuvent être moins susceptibles de provoquer des effets néfastes hors cible et peuvent être inversées. Elles peuvent également être plus subtiles, augmentant ou diminuant légèrement l'activité d'un gène, plutôt que de le faire exploser à pleine puissance ou de l'effacer complètement. "Ce qui est excitant, c'est qu'il y a tellement de choses différentes que vous pouvez faire avec la technologie", explique Charles Gersbach, chercheur de longue date en édition d'épigénomes à l'Université Duke.
L'ajout ou la suppression des étiquettes chimiques sur l'ADN et les protéines histones autour desquelles il s'enroule peut soit étouffer un gène, soit exposer sa séquence de bases d'ADN à d'autres protéines qui l'activent. Certains médicaments anticancéreux enlèvent ou ajoutent ces étiquettes chimiques, mais en tant que combattants de la maladie, ils ont eu un succès limité. L'un des problèmes est que les médicaments ne sont pas ciblés, agissant sur de nombreux gènes à la fois, pas seulement sur ceux liés au cancer, ce qui signifie qu'ils s'accompagnent d'effets secondaires toxiques.
Mais l'édition de l'épigénome peut être rendue précise en exploitant les mêmes enzymes que les cellules utilisent pour activer et désactiver leurs gènes. Les chercheurs attachent des composants clés de ces protéines à une protéine d'édition de gènes, telle qu'une version "morte" de la protéine Cas9 de CRISPR, capable de se diriger vers un endroit spécifique du génome mais incapable de couper l'ADN. Leurs effets peuvent varier : un éditeur peut supprimer les étiquettes des histones pour activer un gène, tandis qu'un autre peut ajouter des groupes méthyle à l'ADN pour le réprimer.
Il y a deux décennies, la société de biotechnologie Sangamo Therapeutics a conçu un éditeur d'épigénome utilisant cette méthode qui a exprimé un gène appelé VEGF, qui aide à favoriser la croissance des vaisseaux sanguins, dans l'espoir de rétablir le flux sanguin chez les personnes atteintes de neuropathie due au diabète. La société a injecté de l'ADN codant pour l'éditeur dans les muscles des jambes d'environ 70 patients lors d'un essai clinique, mais le traitement n'a pas très bien fonctionné. "Nous ne pouvions pas le délivrer efficacement" aux tissus musculaires, déclare Fyodor Urnov, un ancien scientifique de Sangamo maintenant à l'Institut de génomique innovante de l'Université de Californie (UC) à Berkeley.
L'entreprise s'est donc tournée vers un virus adéno-associé (AAV), un virus inoffensif utilisé depuis longtemps en thérapie génique pour délivrer efficacement l'ADN aux cellules. La machinerie de fabrication de protéines de la cellule, pensait-on, utiliserait l'ADN codant pour un éditeur d'épigénome pour en faire un approvisionnement régulier. Cette stratégie semble plus prometteuse : au cours des 3 dernières années, Sangamo a rapporté que chez la souris, il peut réduire les niveaux cérébraux de la protéine tau, une protéine impliquée dans la maladie d'Alzheimer, ainsi que les niveaux de la protéine qui cause la maladie de Huntington.
D'autres équipes travaillant avec des souris utilisent l'approche d'administration d'AAV pour augmenter des niveaux anormalement bas d'une protéine afin de traiter une forme héréditaire d'obésité, ainsi que le syndrome de Dravet, une forme grave d'épilepsie. L'année dernière, un groupe a utilisé l'édition de l'épigénome pour désactiver un gène impliqué dans la perception de la douleur pendant des mois, une alternative potentielle aux médicaments de type opioïdes. Une autre équipe a récemment activé un gène avec un éditeur d'épigénome délivré par un virus différent de l'AAV. Ils l'ont injecté à de jeunes rats exposés à l'alcool ; l'alcool étouffait l'activité d'un gène, ce qui rendait les animaux anxieux et enclins à boire. L'éditeur d'épigénome a réveillé le gène et soulagé les symptômes, a rapporté l'équipe en mai dans Science Advances.
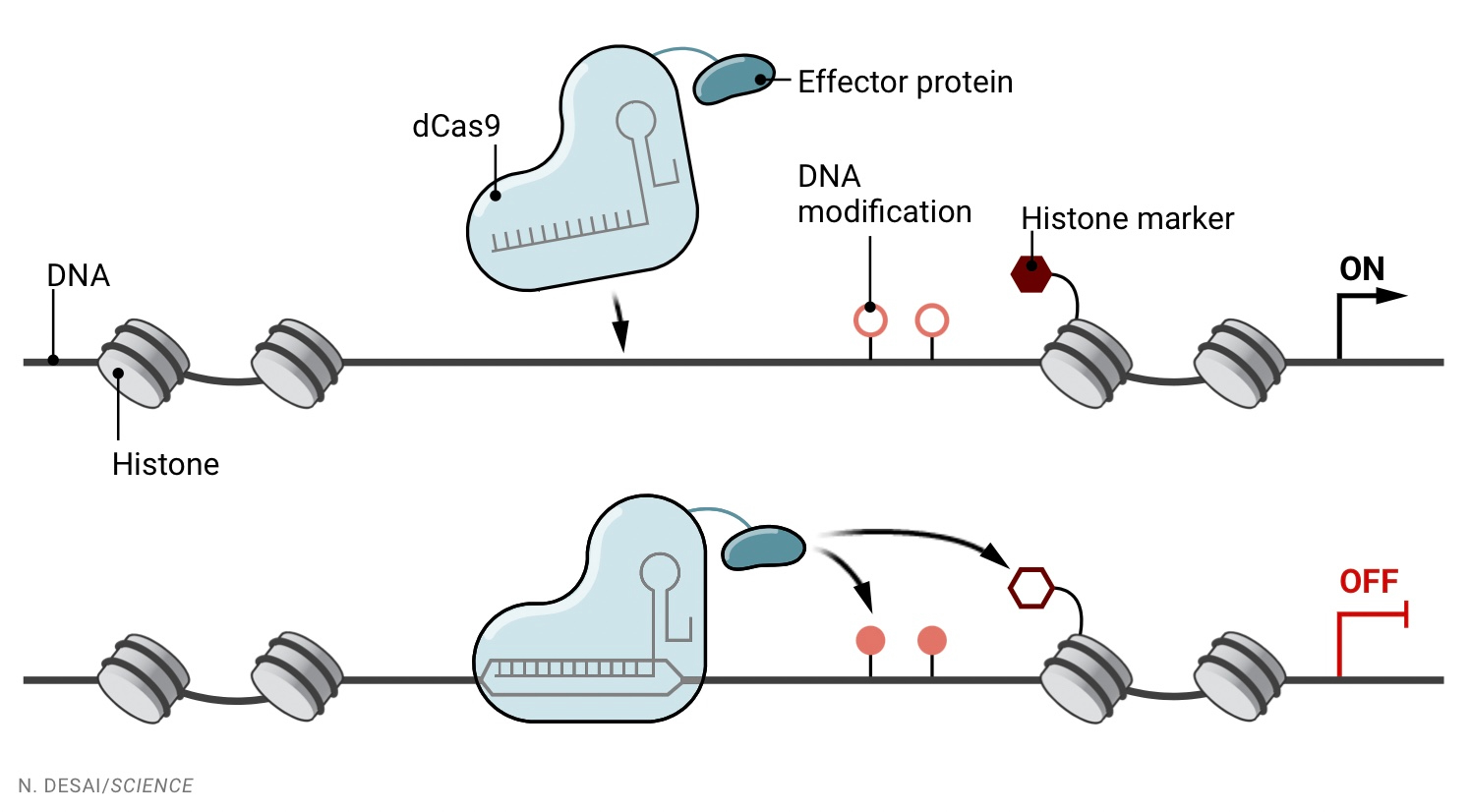
Ensuite, une protéine «effectrice» attachée ajoute ou supprime des étiquettes chimiques sur l'ADN et les protéines histones
autour desquelles elle s'enroule, augmentant ou diminuant l'activité des gènes.
Les AAV testés par de nombreux groupes sont coûteux, et ces porteurs d'ADN, ainsi que les protéines étrangères qu'ils codent, peuvent déclencher une réponse immunitaire. Un autre inconvénient est que la boucle d'ADN codant pour l'éditeur d'épigénome est progressivement perdue dans les cellules lorsqu'elles se divisent.
Le mois dernier, lors de la réunion annuelle de l'American Society of Gene and Cell Therapy à Washington, D.C., des experts en édition de gènes ont proposé une alternative pour éviter les inconvénients des AAV. Une étape clé pour le groupe, dirigé par Angelo Lombardo à l’Institut Téléthon de San Raffaele pour la thérapie génique, est survenue en 2016, lorsque lui, Luigi Naldini et d'autres ont rapporté dans Cell que l'ajout d'un cocktail de trois éditeurs d'épigénome différents aux cellules dans une boite de pétri a réprimé l'expression des gènes et que cela a continué pendant que les cellules se divisaient.
Cela signifiait qu'au lieu de compter sur les AAV pour transporter l'ADN de leurs éditeurs d'épigénome - et forcer l'expression sans fin - ils pouvaient utiliser des nanoparticules lipidiques, une sorte de bulle de graisse, pour porter l’empreinte de l’ADN de l’éditeur d’épigénome, en tant qu'ARN messager (ARNm). De cette façon, les cellules ne fabriquent la protéine que pendant une courte période, ce qui est moins susceptible de déclencher une réponse immunitaire ou de modifier l'épigénome dans des endroits non souhaités. Ces nanoparticules sont largement considérées comme sûres, en particulier après avoir été injectées à des centaines de millions de personnes au cours des 2 dernières années pour délivrer l'ARNm des vaccins COVID-19.
Il a fallu encore plusieurs années à l'équipe italienne pour convertir son étude en laboratoire en succès chez un animal. Lors de la réunion de génomique, le postdoc Martino Cappelluti du laboratoire de Lombardo a expliqué comment l'équipe a injecté à des souris des particules de graisse portant des ARNm codant pour des éditeurs d'épigénome conçus pour faire taire un gène vivant, PCSK9, qui influence le taux de cholestérol. La stratégie a fonctionné, avec une injection supprimant les taux sanguins de la protéine PCSK9 de 50% et réduisant les lipoprotéines de basse densité, ou «mauvais», cholestérol pendant au moins 180 jours.
"Je vois cela comme une formidable avancée", déclare Urnov, qui espère que l'approche des nanoparticules lipidiques sera bientôt étendue à d'autres gènes pathologiques. "L'essentiel ici est que vous n'avez pas besoin d'avoir une expression continue de l'éditeur d'épigénome", explique Jonathan Weissman du Whitehead Institute. Weissman a codirigé des travaux publiés l'année dernière dans Cell sur des éditeurs d'épigénome améliorés basés sur CRISPR qui apportent des changements durables.
Les chercheurs disent que l'édition de l'épigénome pourrait être particulièrement utile pour contrôler plus d'un gène, ce qui est plus difficile à faire en toute sécurité avec l'édition de l'ADN. Cela pourrait traiter des maladies comme le syndrome de Dravet où une personne fabrique une partie d'une protéine nécessaire mais pas assez, car comme un dimmer de lumière, la stratégie peut moduler l'expression des gènes sans l'activer ou la désactiver complètement. Plusieurs nouvelles entreprises espèrent commercialiser des traitements utilisant des éditeurs d'épigénomes. (Gersbach et Urnov en ont fondé une, Tune Therapeutics ; Lombardo, Naldini et Weissman sont parmi les fondateurs d'une autre, Chroma Medicine.)
Malgré l'excitation, les chercheurs préviennent qu'il faudra du temps pour que l'édition de l'épigénome ait un large impact. Les éditeurs ne fonctionnent pas toujours comme annoncé sur certains gènes, explique David Segal, chercheur en épigénétique à l'UC Davis. C'est peut-être en partie parce que, comme s’en inquiète le chercheur en épigénétique John Stamatoyannopoulos de l'Université de Washington, Seattle, les chercheurs ne comprennent pas exactement ce que font les éditeurs une fois qu'ils infiltrent les cellules. "C'est une boîte noire", dit-il.
Pourtant, Stamatoyannopoulos convient que l'édition de l'épigénome est "une formidable promesse". Désormais, les chercheurs doivent affiner leurs éditeurs d'épigénomes, les essayer sur d'autres gènes et tissus pathologiques et les tester sur des animaux plus gros pour des raisons de sécurité avant de passer aux humains.